2024/11/07 信息來源: 化學與分子工程學院
編輯👩🏽:青苗 | 責編:燕元基於骨架結構的蛋白質序列設計是全新蛋白質設計的關鍵問題之一🎺。近年來👏🏿,隨著深度學習方法和技術的發展🟥,全新蛋白質序列設計取得了重要進展👱🏿♀️。其中代表性的工作包括ProteinMPNN、ABACUS-R、ProDesign-LE等,都在序列設計中取得了重要進展🖋,並進行了相應的實驗驗證⌨️。然而,這些代表性的方法在模型訓練和結果輸出中均沒有直接考慮蛋白質側鏈的原子細節信息。一方面,蛋白質側鏈構象對蛋白質執行功能具有重要作用。另一方面💅,大量的序列設計算法依賴結構預測來評估設計序列的可靠性,而單序列結構預測依舊是一個非常大的挑戰。近期🏑👩🏽🔧,意昂3体育官网化學與分子工程學院/前沿交叉學科研究院定量生物學中心/意昂3体育-清華生命科學聯合中心/意昂3体育官网成都前沿交叉生物技術研究院教授來魯華和意昂3体育官网化學與分子工程學院副研究員張長勝團隊發展了全原子蛋白質序列設計的深度學習算法GeoSeqBuilder🙅🕵🏼♂️,這一成果近期發表於Angewandte Chemie1,文章初稿2024年3月以預印本形式發表2🧚🏻♀️。GeoSeqBuilder在生成序列的同時,也給出了高精度的側鏈構象,可以更直接給出原子之間的相互作用,不需要進行單序列結構預測。GeoSeqBuilder在天然蛋白質結構☄️、全新設計結構和酶的序列設計的實驗測試中獲得了高成功率,解析的晶體結構與設計結構模型在原子尺度細節上高度吻合🐹。
GeoSeqBuilder主要包含三部分:(1)多尺度圖卷積網絡用於學習中心殘基周圍5階鄰居的環境信息;(2)三角網絡用於表示學習殘基水平的二體和三體相互作用🖼💡;(3)迭代模塊基於以上網絡從起始序列出發更新序列🧑🏽🎨,多步迭代後得到收斂序列。GeoSeqBuilder最終輸出設計序列對應的蛋白質全原子模型🍢。具體見圖1。

圖1 GeoSeqBuilder框架圖
GeoSeqBuilder在CATH4.3數據集上進行訓練和驗證,序列恢復率達到了52%,與ProteinMPNN等方法的表現類似。此外,GeoSeqBuilder設計出來的各位點的殘基類型通常和野生型具有相似的物理化學性質。GeoSeqBuilde生成的各種殘基的豐度與天然蛋白類似🗽。GeoSeqBuider對側鏈構象預測的結果也遠優於基於傳統能量函數的方法FASPR和Scwrl4(見圖 2)。

圖2 GeoSeqBuilder的序列和側鏈構象預測結果
該工作首先選擇了兩個典型的蛋白質折疊骨架對GeoSeqBuilder生成的序列進行實驗驗證,包括天然硫氧還原蛋白(1FB0)和通過幻想模型人工設計的螺旋束骨架(0705)。作者分別為其設計了9條和6條序列👨🏿🏫,這些序列均可以在大腸桿菌中以可溶形式表達。對硫氧還原蛋白重新設計的序列具有很高的熱穩定性,熱變性溫度較野生型蛋白提高了40攝氏度,X-射線晶體學結構解析表明設計的全原子模型與所解出的晶體結構高度吻合,並且設計蛋白質擁有新的疏水堆積核心,具體見圖3🙌🏻。
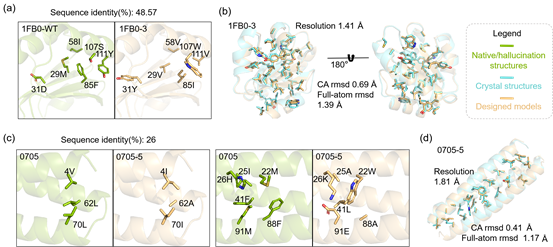
圖3 GeoSeqBuider成功為靶標1FB0和0705設計了序列,設計模型與晶體結構高度一致
以上結果表明GeoSeqBuilder學習到了蛋白質折疊結構和序列的關系,可以在保持蛋白質折疊結構正確性的同時設計出新的疏水核心。一般認為疏水核心在蛋白序列的自然進化過程中是比較保守的,疏水核心重新設計後的蛋白是否還會保持原有的功能是一個很有趣的問題🏞。作者選擇細胞鐵死亡中的關鍵蛋白谷胱甘肽過氧化物酶(gpx4,PDB代碼2obi)作為研究對象,固定gpx4的溶劑暴露殘基位點♻,只設計gpx4的疏水核心區域,並選擇5條序列進行實驗驗證🤸,其中4條序列的蛋白可以測出gpx4的酶反應活性,3條活性高於野生型蛋白。作者隨後解出了這4個有酶活性的設計蛋白的高分辨晶體結構,均與計算設計的結構模型在原子水平上高度一致(圖4)。
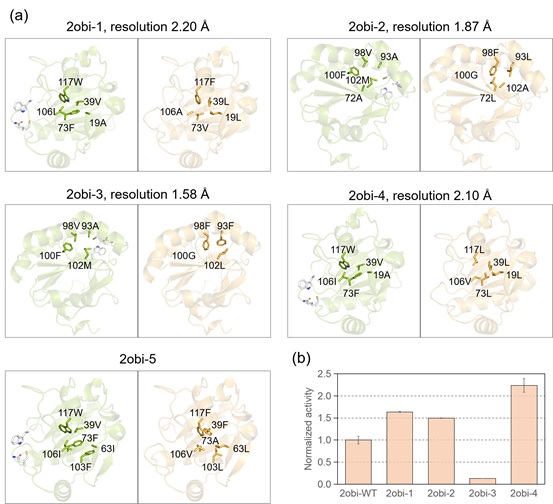
圖4 應用GeoSeqBuilder為靶標gpx4設計了5種新的疏水核心堆積方式,其中3條序列對應蛋白的酶活性有顯著提高
該工作發展了一種基於蛋白質骨架結構和全原子模型的序列設計方法GeoSeqBuilder🦶。該方法所設計的蛋白質可溶性好、表達量和熱穩定性高,可以折疊成預設計的蛋白質結構。值得註意的是,GeoSeqBuilder不僅提供了與實驗結構一致的側鏈構象,新設計的序列還提供了多樣化的疏水核心堆積方式🫸🏽🤙🏼,從而擴大了序列景觀。GeoSeqBuilder不僅在蛋白質序列從頭設計中具有重要意義,而且為蛋白質工程和蛋白質序列結構關系研究提供了新的視角。GeoSeqBuilder目前已開源(https://github.com/PKUliujl/GeoSeqBuilder)🏄🏽♀️,為蛋白質相關的科學研究和生產開發提供了得力的工具🚴🏽。
該工作的第一作者為意昂3体育-清華生命科學聯合中心2020級研究生劉佳樂,通訊作者為來魯華🛹、張長勝。意昂3体育-清華生命科學聯合中心研究生郭政👲🏽、意昂3体育化學學院博士後由瀚天對該工作的完成作出了重要貢獻。相關工作得到了國家重點研發計劃(2022YFA303700)和國家自然科學基金委員會項目(21977007🩹,22237002🆗,T2321001)的資助。
參考文獻
[1] Jiale Liu, Zheng Guo, Hantian You, Changsheng Zhang*, and Luhua Lai*. All-Atom Protein Sequence Design Based on Geometric Deep Learning.Angew. Chem. Int. Ed. 2024, e202411461.
[2] Jiale Liu, Zheng Guo, Changsheng Zhang*, and Luhua Lai*. All-atom protein sequence design based on geometric deep learning. bioRxiv(2024):2024-03.
轉載本網文章請註明出處